Junio 2024
Por: Alejandro Hernández
Director de Biotecnología para Centroamérica y el Caribe
ahernandez@croplifela.org
Ya pasaron más de 10 años del descubrimiento de CRISPR [1], lo que pasó y lo que se presagia que pasará parece ciencia ficción, pero no lo es.
En el 2012 se describió por primera vez una herramienta programable y de altísima precisión para la edición del material genético llamada CRISPR-Cas9. El artículo histórico fue publicado en la revista Science el 28 de junio del 2012 [1] y recibió el premio nobel de Química en el 2020 [2]. Este tipo de herramientas de mutación dirigida existían ya en versiones de meganucleasas, TALENS, Nucleasas de dedos de Zinc (ZFNs) y Mutación dirigida con Oligonucleótidos (ODM), pero con mayor costo, dificultad de diseño y precisión en comparación con las herramientas de CRISPR-Cas9 [3], [4]. La comunidad científica y el mundo quedó maravillado por la posibilidad de curar enfermedades hereditarias humanas hasta ahora de difícil abordaje clínico como la betatalasemia, o nuevas terapias para tratar VIH, papiloma o cáncer [5]. En los inicios, era difícil imaginar la importancia de estas herramientas de precisión genética para el sector agropecuario pero su importancia es cada vez más clara por los retos y oportunidades que existen en el sector. Las preguntas que estaremos resolviendo son: (1) qué pasó en estos años, (2) qué logramos avanzar hacia tratamientos y mejoras en la vida humana, el agro y el ambiente, y (3) que será en la siguiente década. Acompáñenos en este artículo para hacer un recorrido hacia lo que ya es pasado, los logros, retos y el futuro de esta tecnología, usando como guía la revisión que hizo la ganadora del Premio Nobel de Química Jenifer Doudna en la revista Science en 2023 [6].
1. Pasado
El primer descubrimiento se basó en encontrar un buscador de material genético, CRISPR-Cas9
Primero un poco de contexto, para los entendidos, esta sección les será repetitivo, pero siempre es bienvenido un poco antecedentes, previo a entrar en materia. CRISPR-Cas9 es una enzima, una proteína que en su versión primitiva es capaz de encontrar material genético complementario a una hebra molde que podemos facilitar al sistema y cortar ese material genético, y por eso en biología se llama nucleasa [7], [8]. La predicción de estructura tridimensional de esta enzima está disponible gracias a Alphafold según se presenta a continuación con el código Uniprot G3ECR1[9] (Figura 1).
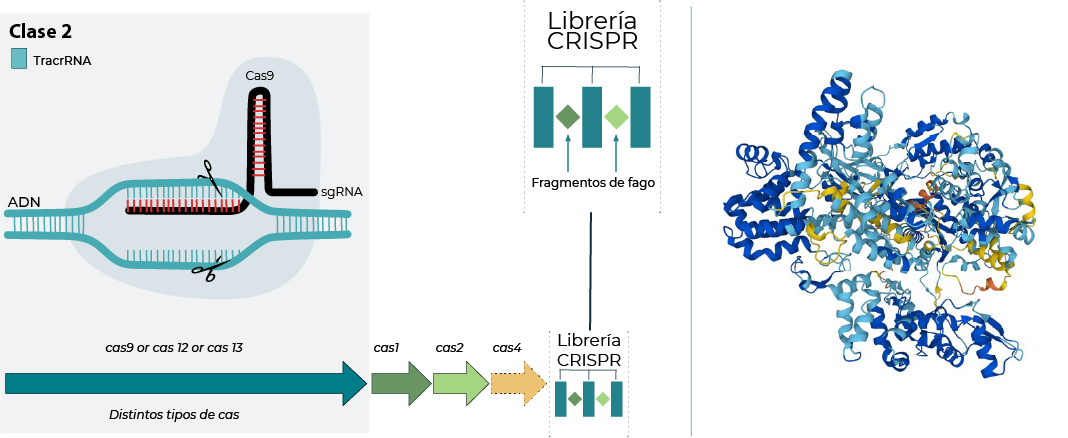
Nótese la estructura tridimensional de la enzima Uniprot G3ECR1
Lo primero que se descubrió no fue la enzima sino los moldes que se unen a la enzima, almacenados en una librería llamada CRISPR (Figura 1). Fue el español Francisco Mojica quien hizo este hallazgo que en ciencia es fascinante porque encontró la librería en un organismo extremófilo en los 90s[10] y que detallaría a inicios del 2000 [11], [12], mientras que una librería similar se había reportado en un procariota por parte de laboratorio del Dr. Nakata a finales de los 80s [13]. Desde el punto de vista evolutivo, la existencia de una librería similar en grupos tan distintos es algo extremadamente infrecuente y daba pistas de su importancia. Años después se descubriría que esta librería era un almacén de defensa, donde se acumulaban de manera ordenada pequeños fragmentos de material genético de fagos, que son los “virus” de los microorganismos. Cada fragmento del fago estaba separado por unas secuencias que se repetían. La librería de fagos y secuencias repetidas en sus bordes, se les llamó CRISPR, que es el término que acuñó Francisco Mojica del acrónimo en inglés “Clustered Regularly Interspaced Short Palindromic Repeats” (CRISPR) (Figura 1).
El segundo descubrimiento fue en cómo usar el buscador de material genético para corregirlo.
Ahora bien, la librería CRISPR es “leída” de alguna manera. Es ahí donde se descubren las enzimas que realizan la función asociada a la librería y se acuñan con el término CAS que es un acrónimo para “CRISPR Associated Proteins” (CAS). En particular la Cas9 es capaz de usar el molde para posteriormente identificar un material genético. En su versión silvestre, la Cas9 utiliza el molde como un mecanismo para identificar a patógenos. El molde es una huella genética única para posteriormente desarticular el virus mediante un corte de la hebra de material genético y por eso es una “nucleasa”. La acción silvestre de cortar el ADN es el motivo por lo que popularmente se la llaman “tijeras moleculares” a las enzimas CRISPR-Cas9.
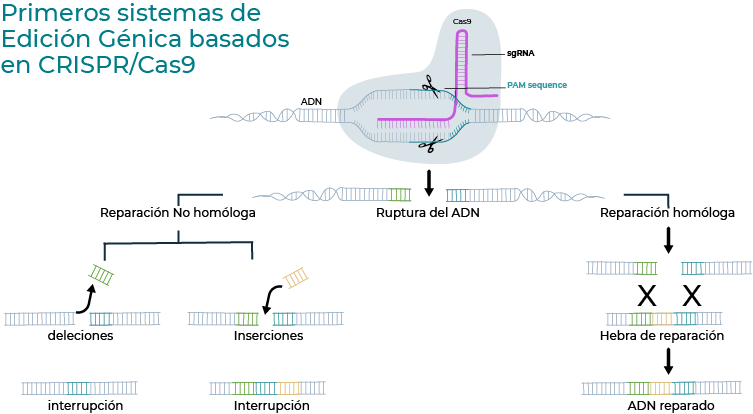
mediante reparación no homóloga o reparación homóloga con una hebra de reparación.
El tercer elemento clave es lograr diseños a la medida para corregir el material genético
La función de edición de genoma no tiene relación alguna con su función primitiva de defensa, sino que se usa el sistema de reparación natural de la célula que es provocado por las enzimas Cas9. El sistema de reparación CRISPR-CAS9 que se planteó en 2012 y por el cual se otorgó el premio nobel de química, consiste en aprovechar la habilidad de identificar el material genético a partir de una hebra molde idéntica al sitio que se desea corregir y aprovechar el sistema natural de reparación del ADN para corregir o inducir un ajuste. La técnica en particular exige de mucho diseño, que en este artículo no abordaré para no saturar al lector con información técnica. Para los curiosos, se requiere conocer el genoma de interés, identificar el error en el material genético de partida, lo que se desea ajustar, la estructura 3D de la hebra, el molde que se diseña para que se acople a la Cas9 llamado protoespaciador, y el sitio en el genoma donde la enzima logra validar el sitio de corte, llamado sitio adjacente al protoespaciador o “PAM”.
Les comparto sitios para diseño para quienes deseen explorar el tema
| SITIO | LINK | REFERENCIA |
| CRISPRdirect | http://crispr.dbcls.jp/ | [14] |
| Cas-Designer | http://www.rgenome.net/cas-designer/ | [15] |
| CRISPOR | http://crispor.tefor.net | [16] |
| CRISPR-P 2.0 | http://crispr.hzau.edu.cn/CRISPR2/ | [17] |
| CHOP-CHOP | https://chopchop.cbu.uib.no | [18] |
2. ¿Qué pasó en esta primera década de CRISPR?.
Se optimizaron las herramientas
La primera década de CRISPR estuvo marcada por un gran desarrollo de las herramientas, optimizando su funcionamiento, efectividad, precisión y diversidad. Se logró identificar enzimas CAS en eucariotas llamadas fanzor y encontrar el ancestro común Omega[19]. La precisión aumentó al punto de tener Cas de alta fidelidad lo que es importantísimo para evitar sitios no blancos en terapias para con una temperatura corporal de 37C y por tanto enzimas más activas en comparación con plantas [20]. Hoy contamos con herramientas para identificar ADN, ARN, realizar mutaciones dirigidas de una sola base que lo logran sin necesidad de cortar del ADN[21], [22], [23]. También es posible reparar una hebra de ADN a partir de un molde de ARN mediante prime Editing[24], [25], [26]. Una mejora significativa es la capacidad de modificar el epigenoma [27] y utilizar múltiples mecanismos para activar o desactivar la expresión génica de manera transitoria. Un avance que demuestra la velocidad con la que avanzan las herramientas es la existencia de enzimas libres de licencia como la MAD7 que es una enzima Cas12a/Cpf1 libre para uso académico y comercial [28]
La herramienta que en algún momento se le llamó tijeras moleculares, ya ni siquiera requieren de cortar el ADN para cumplir con su labor de ajuste dirigido del material genético y son aún más versátiles, precisas y diversas en comparación con sus versiones silvestres.
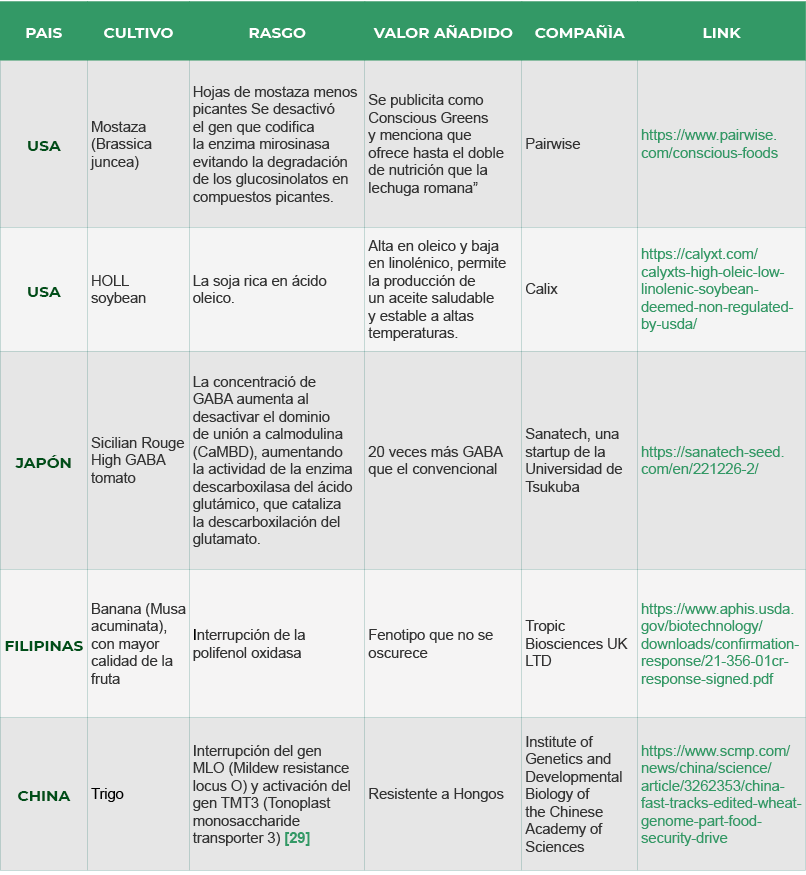
Aparecen ejemplos de cultivos comerciales
Existen ejemplos de cultivos comerciales que tienen un gran avance desde el punto de vista comercial tales como mostaza menos picante, soya rica en ácido oleico, tomate rico en GABA para la presión arterial y banano que no se oscurece, y recientemente en 2024 China sorprendió al autorizar trigo editado resistente al ataque de hongos.
Se aprueba la primer terapias en humanos
En diciembre del 2023 la FDA aprobó una terapia basada en CRISPR para el tratamiento de la Beta talasemia, mismo tratamiento que recibió posteriormente el aval en el Reino Unido, así como la Comunidad Económica Europea en 2024. En junio del 2024 ensayos clínicos en China ChiCTR2200063181 demostraron el uso de CRISPR para corregir una mutación en el gen DFNB9 en los cinco pacientes tratados [30].
Se aprueba animales y se avanza en los modelos con fines terapéuticos
En 2021 Japón autorizó para su uso y consumo dos tipos de peces que crecen con mayor velocidad y desarrollados gracias al apoyo de la Universidad de Kyoto y la Universidad de Kindai [31]. Se espera que para final de este 2024 la FDA autorice los primeros cerdos editados resistentes a virus del síndrome respiratorio y reproductivo porcino (PRRS)[32], [33]. Los modelos de animales para transplantes de órganos son también posibles gracias a CRISPR, en 2024 se realizó el primer transplante de hígado proveniente de un cerdo editado en el Hospital General de Masachusetts, y las investigaciones alrededor de este tema continúan creando modelos en animales [34], [35].
Este antecedente marca un hito en el acceso de tecnologías para terapias humanas y animales, y un futuro prometedor para la transferencia de estos desarrollos al sector agro.
3. Qué sigue?
Evidentemente estamos ante un panorama de mayor exigencia para el sector productivo para adaptar la agricultura al cambio climático, tema que habíamos abordado previamente, ver Cómo adaptar la agricultura al cada vez más evidente cambio climático . En este contexto, el Foro Económico Mundial prevé que CRISPR hará cambios para un mundo mejor en temas de salud y alimentación en cinco puntos. El primero en diagnóstico y prevención de enfermedades, alimentos más nutritivos y resistentes, animales de granja con mejoras en su salud, cultivos resilientes al cambio climático y tratamiento del cáncer.
Avances en desafíos difíciles para la agricultura
Algunos de los rasgos más prometedores están relacionados con grandes retos que enfrenta la agricultura y para los cuales existen muy limitadas opciones. Ejemplos de estos retos son el banano tolerante a Fusarium Raza 4, arroz con capacidad de crecer en agua con alta salinidad oceánica, Caña de azúcar de nueva generación para la producción de bioetanol (BAHD01, BAHD05) de Embrapa [37], cítricos tolerantes a HLB liderados por Yale y la Universidad de Florida, palma aceitera con rasgos interesantes como enanos, bajos en lipasa, mejora en color y resistencia, así como mejora en la calidad de aceite [38], cacao que no acumula cadmio. Otros desarrollos innovadores está relacionados a microorganismos fijadores de nitrógeno o levaduras. La cantidad de desarrollos va en aumento dadas las posibilidades que emergerán en el uso de la tecnología.
Mejoras regulatorias
En el sector agrícola es interesante notar la enorme cantidad de desarrollos sometidos ante el APHIS en Estados Unidos que cuenta con más de 200 distintos fenotipos, la base de datos oficial de la Comunidad Económica Europea del Join Research Centre [36], así como el Europea Eusage que detalla investigaciones que compila más de 800 investigaciones principalmente en arroz, tomate, maíz, soya, trigo, papa, canola y cebada. Todos ellos hacen énfasis en cultivos que sean más resistentes, nutritivos, con valor añadido y más rendimiento.
.
| SITIO | LINK |
| Aphis, USA | https://www.aphis.usda.gov/biotech-exemptions/confirmation-letters |
| JRC | https://datam.jrc.ec.europa.eu/datam/mashup/NEW_GENOMIC_TECHNIQUES/index.html |
| EU sage | https://www.eu-sage.eu/genome |
| Gene Editing Regulation Tracker | https://crispr-gene-editing-regs-tracker.geneticliteracyproject.org |
El continente Américano dispone de una gran avance desde el punto de vista regulatorio. Centroamérica dispone de un modelo sumamente directo que aprovecha el entendimiento jurídico internacional, así como el dado en países como Brasil, Argentina, Paraguay, Colombia, Estados Unidos, y que dispone de decisiones favorables para cultivos de interés para la región como banano de larga vida en anaquel, no pardeamiento y resistencia a hongos.
Avance en cultivos huérfanos y conocimiento de los cultivos
La investigación pública estará generando innovaciones que serán cada vez más importantes en cultivos huérfanos que requieren de mejoramiento para que soporten el cambio climático, aumenten su rendimiento o toleren plagas [39]. La investigación de la genética de los cultivos será cada vez más importante dado que es la base para entender rasgos de interés agronómico que puedan ser trasladados a cultivos comerciales que pueden usar edición de genomas para ganar tiempo para desarrollar variedades cada vez más adaptadas. Un ejemplo de esto es la biodiversidad re-descubierta por el Instituto John Innes analizó las semillas de trigo de los años 1920-1930s de más de 1000 materiales. Tener acceso a lo que se conoce como banco de genes de un cultivo (pool de genes) podría ser clave en el futuro para adaptar mejor los cultivos.
La ganadora del premio nobel nos hace un resumen del futuro en la siguiente imagen [6] donde se presagian desarrollos importantes para la humanidad en la siguiente década. Las mejoras en cultivos, animales serían claves para el agro en la búsqueda de mejoras en el rendimiento, calidad, nutrición y resiliencia al cambio climático.
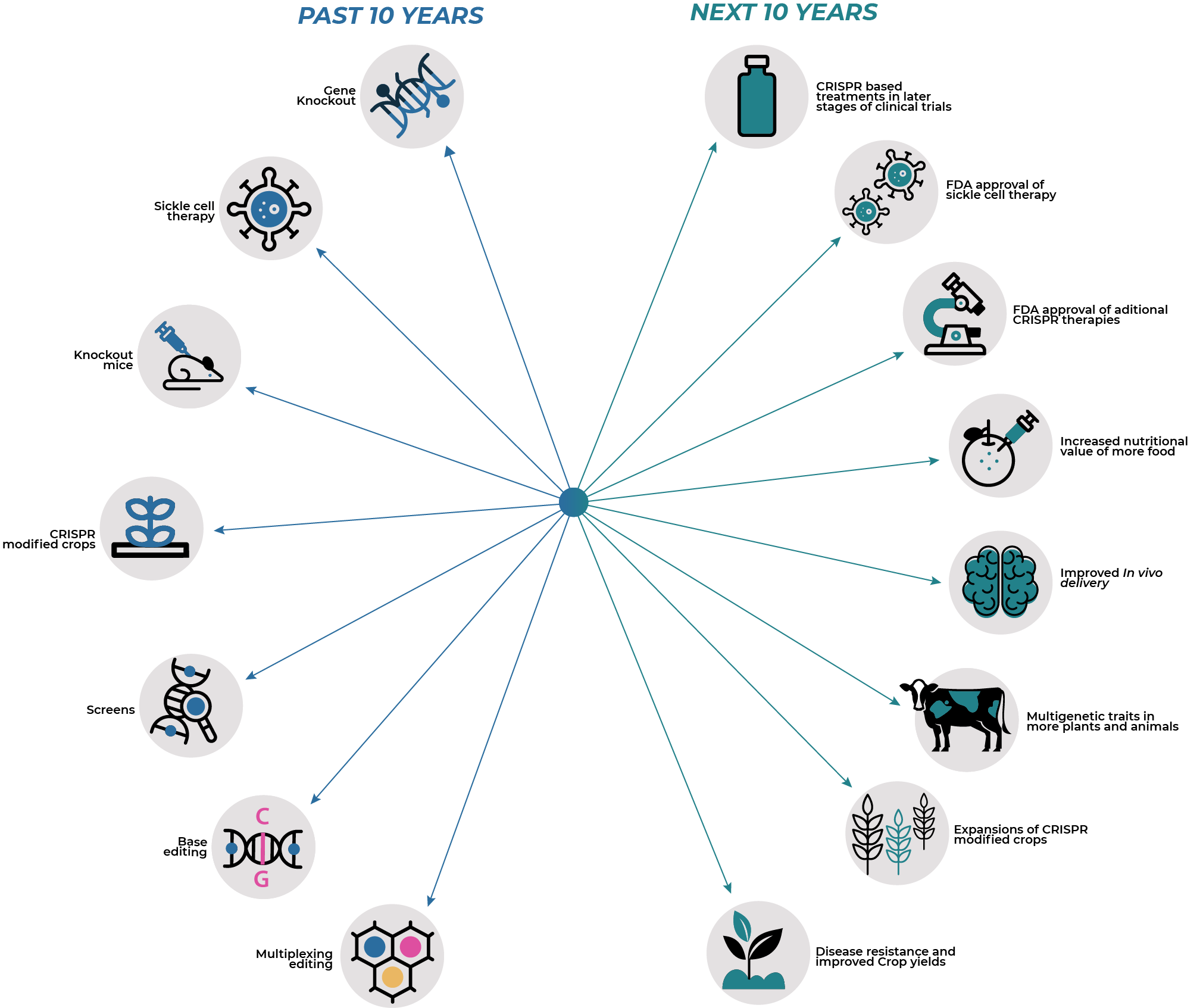
Terapias en humanos de nueva generación
En el caso de terapias humanas, es interesante notar los ensayos clínicos que están en proceso tales como CRISPR para el tratamiento de Cáncer riñón mediante CD70-CAR-T cells con una efectividad del 77%, leucemia con células editadas CAR-T, eliminación del VIH mediado por CRISPR-Cas9 mediante la entrega con adeno-virus AAV9 y la disminución del colesterol mediada por edición en una base para hacer un knock out (KO) del gen PCSK9.
Integración con nuevas tecnologías
El futuro es muy interesante no solamente por las herramientas como CRISPR, sino porque confluyen una serie de elementos que en el pasado estaban ausentes. Ejemplos de estas herramientas son el acceso a una secuenciación de genomas a muy bajo costo y la inteligencia artificial que permite crear modelos a partir de bases de datos relacionados al genotipo y al fenotipo. Lo que el ser humano logró basado en seleccionar alimentos y que le costó miles de años, hoy es posible de replicar, aprender, recrear y dar rasgos a cultivos actuales o nuevos usando la misma lógica, pero en mucho menor tiempo. El mundo de la investigación pública y privada mira con mucho orgullo lo que pasó en esta década de CRISPR y lo que se presagia que pasará en la siguiente década parece ciencia ficción, pero no lo es.
Bibliografía citada
[1] M. Jinek, K. Chylinski, I. Fonfara, M. Hauer, J. A. Doudna, and E. Charpentier, “A Programmable Dual-RNA–Guided DNA Endonuclease in Adaptive Bacterial Immunity,” Science (1979), vol. 337, no. 6096, pp. 816–821, Aug. 2012, doi: 10.1126/science.1225829.[2] A. Fernholm and C. Barnes, “The Nobel Prize in Chemistry 2020 Genetic scissors: a tool for rewriting the code of life,” The Royal Swedish Academy of Science, p. 8, 2020, [Online]. Available: https://www.nobelprize.org/prizes/chemistry/2020/popular-information/[3] M. Kashtwari et al., “Random mutagenesis in vegetatively propagated crops: opportunities, challenges and genome editing prospects,” Mol Biol Rep, 2021, doi: 10.1007/s11033-021-06650-0.[4] D. D. Songstad, J. F. Petolino, D. F. Voytas, and N. A. Reichert, “Genome Editing of Plants,” CRC Crit Rev Plant Sci, vol. 36, no. 1, pp. 1–23, Jan. 2017, doi: 10.1080/07352689.2017.1281663.[5] F. Morshedzadeh et al., “An Update on the Application of CRISPR Technology in Clinical Practice,” Mol Biotechnol, vol. 66, no. 2, pp. 179–197, 2024, doi: 10.1007/s12033-023-00724-z.[6] J. Y. Wang and J. A. Doudna, “CRISPR technology: A decade of genome editing is only the beginning,” Science (1979), vol. 379, no. 6629, Jan. 2023, doi: 10.1126/science.add8643.[7] J. A. Doudna and E. Charpentier, “The new frontier of genome engineering with CRISPR-Cas9,” Science (1979), vol. 346, no. 6213, Nov. 2014, doi: 10.1126/science.1258096.[8] H. Wang, M. La Russa, and L. S. Qi, “CRISPR/Cas9 in Genome Editing and Beyond,” Annu Rev Biochem, vol. 85, no. 1, pp. 227–264, 2016, doi: 10.1146/annurev-biochem-060815-014607.[9] J. Jumper et al., “Highly accurate protein structure prediction with AlphaFold,” Nature, vol. 596, no. 7873, pp. 583–589, 2021, doi: 10.1038/s41586-021-03819-2.[10] F. J. M. Mojica, G. Juez, and F. Rodriguez‐Valera, “Transcription at different salinities of Haloferax mediterranei sequences adjacent to partially modified Pst I sites,” Mol Microbiol, vol. 9, no. 3, pp. 613–621, Aug. 1993, doi: 10.1111/j.1365-2958.1993.tb01721.x.[11] F. J. M. Mojica, C. Díez-Villaseñor, J. García-Martínez, and E. Soria, “Intervening Sequences of Regularly Spaced Prokaryotic Repeats Derive from Foreign Genetic Elements,” J Mol Evol, vol. 60, no. 2, pp. 174–182, Feb. 2005, doi: 10.1007/s00239-004-0046-3.[12] F. J. M. Mojica, C. Díez‐Villaseñor, E. Soria, and G. Juez, “Biological significance of a family of regularly spaced repeats in the genomes of Archaea, Bacteria and mitochondria,” Mol Microbiol, vol. 36, no. 1, pp. 244–246, Apr. 2000, doi: 10.1046/j.1365-2958.2000.01838.x.[13] Y. Ishino, H. Shinagawa, K. Makino, M. Amemura, and A. Nakata, “Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product,” J Bacteriol, vol. 169, no. 12, pp. 5429–5433, Dec. 1987, doi: 10.1128/jb.169.12.5429-5433.1987.
[14] Y. Naito, K. Hino, H. Bono, and K. Ui-Tei, “CRISPRdirect: Software for designing CRISPR/Cas guide RNA with reduced off-target sites,” Bioinformatics, vol. 31, no. 7, pp. 1120–1123, 2015, doi: 10.1093/bioinformatics/btu743.
[15] J. Park, S. Bae, and J. S. Kim, “Cas-Designer: A web-based tool for choice of CRISPR-Cas9 target sites,” Bioinformatics, vol. 31, no. 24, pp. 4014–4016, 2015, doi: 10.1093/bioinformatics/btv537.
[16] J. P. Concordet and M. Haeussler, “CRISPOR: Intuitive guide selection for CRISPR/Cas9 genome editing experiments and screens,” Nucleic Acids Res, vol. 46, no. W1, pp. W242–W245, 2018, doi: 10.1093/nar/gky354.
[17] Y. Lei, L. Lu, H. Y. Liu, S. Li, F. Xing, and L. L. Chen, “CRISPR-P: A web tool for synthetic single-guide RNA design of CRISPR-system in plants,” Mol Plant, vol. 7, no. 9, pp. 1494–1496, 2014, doi: 10.1093/mp/ssu044.
[18] K. Labun, T. G. Montague, M. Krause, Y. N. Torres Cleuren, H. Tjeldnes, and E. Valen, “CHOPCHOP v3: Expanding the CRISPR web toolbox beyond genome editing,” Nucleic Acids Res, vol. 47, no. W1, pp. W171–W174, 2019, doi: 10.1093/nar/gkz365.
[19] M. Saito et al., “Fanzor is a eukaryotic programmable RNA-guided endonuclease,” Nature, vol. 620, no. 7974, pp. 660–668, 2023, doi: 10.1038/s41586-023-06356-2.
[20] R. Rabinowitz and D. Offen, “Single-Base Resolution: Increasing the Specificity of the CRISPR-Cas System in Gene Editing,” Molecular Therapy, vol. 29, no. 3, pp. 937–948, 2021, doi: 10.1016/j.ymthe.2020.11.009.
[21] C. Kuscu et al., “CRISPR-STOP: Gene silencing through base-editing-induced nonsense mutations,” Nat Methods, vol. 14, no. 7, pp. 710–712, 2017, doi: 10.1038/nmeth.4327.
[22] V. Marx, “Base editing a CRISPR way,” Nat Methods, vol. 15, no. 10, pp. 767–770, 2018, doi: 10.1038/s41592-018-0146-4.
[23] C. Li et al., “Targeted, random mutagenesis of plant genes with dual cytosine and adenine base editors,” Nat Biotechnol, vol. 38, no. 7, pp. 875–882, 2020, doi: 10.1038/s41587-019-0393-7.
[24] Q. Lin et al., “Prime genome editing in rice and wheat,” Nat Biotechnol, vol. 38, no. 5, pp. 582–585, 2020, doi: 10.1038/s41587-020-0455-x.
[25] W. Xu et al., “Versatile Nucleotides Substitution in Plant Using an Improved Prime Editing System,” Mol Plant, vol. 13, no. 5, pp. 675–678, 2020, doi: 10.1016/j.molp.2020.03.012.
[26] H. Li, J. Li, J. Chen, L. Yan, and L. Xia, “Precise Modifications of Both Exogenous and Endogenous Genes in Rice by Prime Editing,” Mol Plant, vol. 13, no. 5, pp. 671–674, 2020, doi: 10.1016/j.molp.2020.03.011.
[27] J. K. Nuñez et al., “Genome-wide programmable transcriptional memory by CRISPR-based epigenome editing,” Cell, vol. 184, no. 9, pp. 2503-2519.e17, 2021, doi: 10.1016/j.cell.2021.03.025.
[28] Q. Lin et al., “Genome editing in plants with MAD7 nuclease,” Journal of Genetics and Genomics, vol. 48, no. 6, pp. 444–451, 2021, doi: 10.1016/j.jgg.2021.04.003.
[29] S. Li et al., “Genome-edited powdery mildew resistance in wheat without growth penalties,” Nature, vol. 602, no. 7897, pp. 455–460, 2022, doi: 10.1038/s41586-022-04395-9.
[30] H. Wang et al., “Bilateral gene therapy in children with autosomal recessive deafness 9: single-arm trial results,” Nat Med, vol. 9, Jun. 2024, doi: 10.1038/s41591-024-03023-5.
[31] “Japan embraces CRISPR-edited fish,” Nat Biotechnol, vol. 40, no. 1, pp. 10–10, Jan. 2022, doi: 10.1038/s41587-021-01197-8.
[32] J. Cohen, “Meat from gene-edited pigs could hit the market,” Science (1979), vol. 383, no. 6686, pp. 940–941, Mar. 2024, doi: 10.1126/science.ado9328.
[33] B. T. Burger et al., “Generation of a Commercial-Scale Founder Population of Porcine Reproductive and Respiratory Syndrome Virus Resistant Pigs Using CRISPR-Cas,” CRISPR Journal, vol. 7, no. 1, pp. 12–28, 2024, doi: 10.1089/crispr.2023.0061.
[34] J. J. Y. Lim, Y. Murata, S. Yuri, K. Kitamuro, T. Kawai, and A. Isotani, “Generating an organ-deficient animal model using a multi-targeted CRISPR-Cas9 system,” Sci Rep, vol. 14, no. 1, pp. 1–12, 2024, doi: 10.1038/s41598-024-61167-3.
[35] R. P. Anand et al., “Design and testing of a humanized porcine donor for xenotransplantation,” Nature, vol. 622, no. 7982, pp. 393–401, 2023, doi: 10.1038/s41586-023-06594-4.
[36] C. Parisi and E. Rodriguez-Cerezo, “Current and future market applications of new genomic techniques,” 2021. doi: 10.2760/02472.
[37] G. S. Prado et al., “CRISPR technology towards genome editing of the perennial and semi-perennial crops citrus, coffee and sugarcane,” Front Plant Sci, vol. 14, no. January, pp. 1–27, Jan. 2024, doi: 10.3389/fpls.2023.1331258.
[38] B. Bahariah, M. Y. A. Masani, M. P. M. A. A. Fizree, O. A. Rasid, and G. K. A. Parveez, “Multiplex CRISPR/Cas9 gene-editing platform in oil palm targeting mutations in EgFAD2 and EgPAT genes,” Journal of Genetic Engineering and Biotechnology, vol. 21, no. 1, p. 3, Dec. 2023, doi: 10.1186/s43141-022-00459-5.
[39] A. Tyczewska, T. Twardowski, and E. Woźniak-Gientka, “Agricultural biotechnology for sustainable food security,” Trends Biotechnol, vol. 41, no. 3, pp. 331–341, Mar. 2023, doi: 10.1016/j.tibtech.2022.12.013.